 当前位置:首页 > 新闻中心 > 新闻资讯
当前位置:首页 > 新闻中心 > 新闻资讯
神经退行性疾病是一种神经系统慢性进行性损害疾病,以远端轴突损伤和逆死性神经病变为主要特征,然而从轴突损伤到神经元死亡的途径仍不完全清楚。基因表达的变化是神经损伤的重要表现,在体内高通量筛选转录因子,以确定神经元损伤或轴突再生的关键调控因子是研究神经退行性疾病的有效方案,CRISPR-gRNA文库筛选结合多组学分析为实现体内大规模遗传筛选提供了双重保障。
文章标题:Core transcription programs controlling injury-induced neurodegeneration of retinal ganglion cells.
发表期刊:Neuron(IF=18.688)
合作客户:哈佛医学院
维真助力:
小鼠gRNA AAV载体库
· 维真生物 ·
小鼠gRNA AAV载体库
【点击查看详情】
种属:小鼠
数量:近13000
载体:AAV
应用:可以直接用于AAV包装,可与spCas9 AAV病毒或者spCas9转基因动物搭配使用,轻松实现目的基因的体内基因敲除,方便目的基因的功能研究。
优势: 1、载体GFP荧光标签,便于监测质粒转染效率;2、可以直接进行AAV包装;3、与单条gRNA相比,每个目的基因4-5条gRNA pool的设计,极大增加敲除效率;4、可提供整个小鼠gRNA pool 克隆库,也可提供各类子库,如转录因子、蛋白激酶、磷酸酶、药物靶点、膜蛋白等。5、全部现货,大大缩短科研周期。
多数神经退行性疾病表现为相同的病理特征,其中极具破坏性的是神经元死亡,但目前神经退行性疾病中神经元死亡和轴突再生的调控程序仍知之甚少。为了确定成年个体中枢神经系统轴突损伤导致神经退行性变的机制,研究人员利用了视神经挤压(ONC)模型,即通过切断视网膜神经节细胞(RGC)轴突导致ONC,进而导致大量RGC死亡和再生失败。基于CRISPR-gRNA文库筛选技术,研究人员对1893个转录因子(TF)进行全基因组筛选,寻找ONC后RGC存活和轴突再生的抑制因子,同时通过ATAC-seq和RNA-seq等分析损伤RGC的表观遗传和转录情况,确定了损伤响应性TF及其靶标。研究结果揭示了从轴突损伤转化为神经退行性疾病的核心转录程序,并提出了治疗神经退行性疾病的新策略。
1. CRISPR-gRNA文库筛选技术结合表观遗传分析筛选神经元存活和轴突再生的负调控TF
作者利用RIKEN和TFCat数据库汇总了1893个小鼠基因组中的TF,并针对每个TF基因不同区域,从CRISPR-Cas9敲除(GeCKO)文库中选择了五条sgRNAs,然后pool在一起克隆至AAV2载体,建立了一个包含1893个AAV2-sgRNA的文库(维真生物助力提供)。随后借助AAV2将sgRNAs和Cas9引入RGC,ONC后两周进行筛选检测以确定调控神经元存活和轴突再生的TF。实验结果确定了10个负调控神经元存活的TF和13个负调控轴突再生的TF。通过整合ATAC-seq,RNA-seq,TF footprinting信息筛选到4种负调控RGCs损伤存活的关键TF:ATF3、ATF4、C/EBPγ和CHOP。之后作者重点探究了这四个关键的TF调控的转录程序,发现ATF3/CHOP优先调节由细胞因子和先天免疫激活的通路,ATF4/C/EBPγ调节由内在神经元应激源激活的通路。
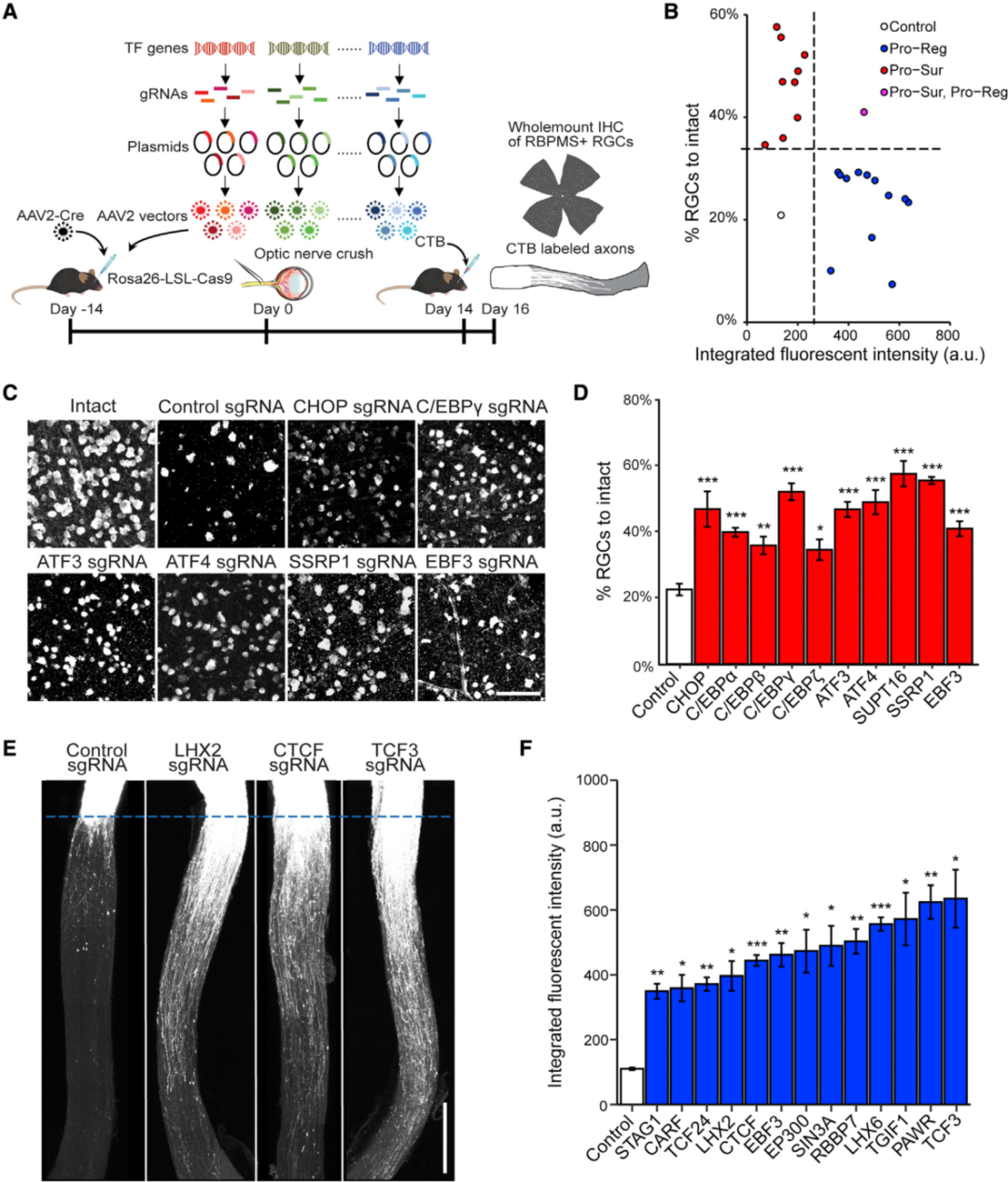
2. 受损RGC中的关键TF存在功能性交互作用
ATF3/CHOP和C/EBPγ/ATF4作为ONC后RGC存活和轴突再生的两组负转录调控因子,作者推测与单个TF敲除相比,联合操纵来自两组的TF可能具有叠加效应,并进一步提高RGC的存活率。作者发现:TF的单个敲除相较于对照组显著提高了RGC的存活率;与敲除单个TF相比,TF的交替组合敲除(ATF3/C/EBPγ, ATF3/ATF4和C/EBPγ/CHOP)显著提高RGC存活率;但同组内共敲除TF(ATF3/CHOP 或 ATF4/C/EBPg)的保护作用并不明显。这表明ATF3/CHOP和ATF4/C/EBPγ在神经元损伤后激活两条平行的促死亡通路,两通路间存在重叠和互补作用。

本研究通过独立的体内遗传筛选和损伤视网膜神经节细胞的多组学分析,确定了四种损伤后神经元存活的关键互补调控因子(ATF3、CHOP、ATF4、C/EBPγ),这些基因分别调节两种不同的退行性程序,以应对外在和内在的压力。
备案号:鲁ICP备13011878号 网址:www.wzbio.com.cn









